亲和力成熟
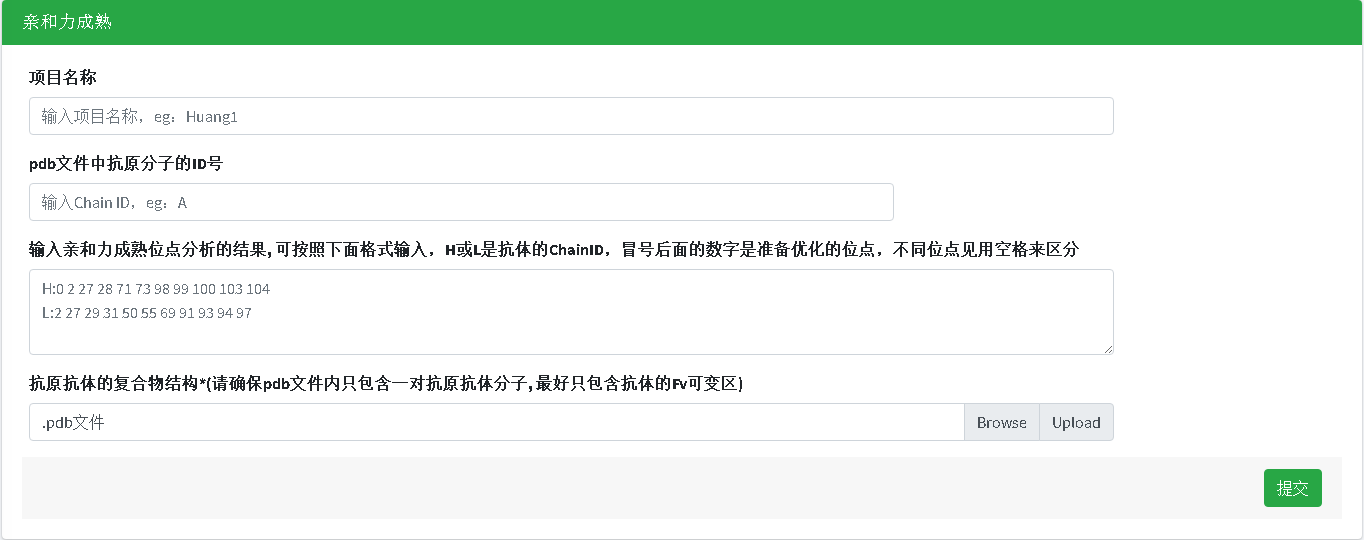
根据上图界面提示,请输入编辑好的项目名、正反向引物序列、压缩的测序结果文件。
项目名称:需要客户根据自己的实际项目来提供一个编写的项目名称。
pdb文件中抗原分子的ID号:因为此项功能需要抗原抗体复合物的晶体结构或者是预测的复合物结构,因此请在此输入抗原抗体的复合物结构中抗原的ID号,如A或B等等来区分抗体与抗原。
输入亲和力成熟位点分析的结果:请在此输入**亲和力成熟位点分析**功能的结果,客户可以在此进行某些位点的增加与删除。
抗原抗体的复合物结构:如上文所述,因为需要抗原抗体结合的晶体结构或者预测的结构,因此请在此输入,最好在结构中的抗体部分只保留Fv可变区。
都输入好后点击提交,静等结果反馈,可以在结果列表里面查看运行状态。当运行结束时会得到如下结果:
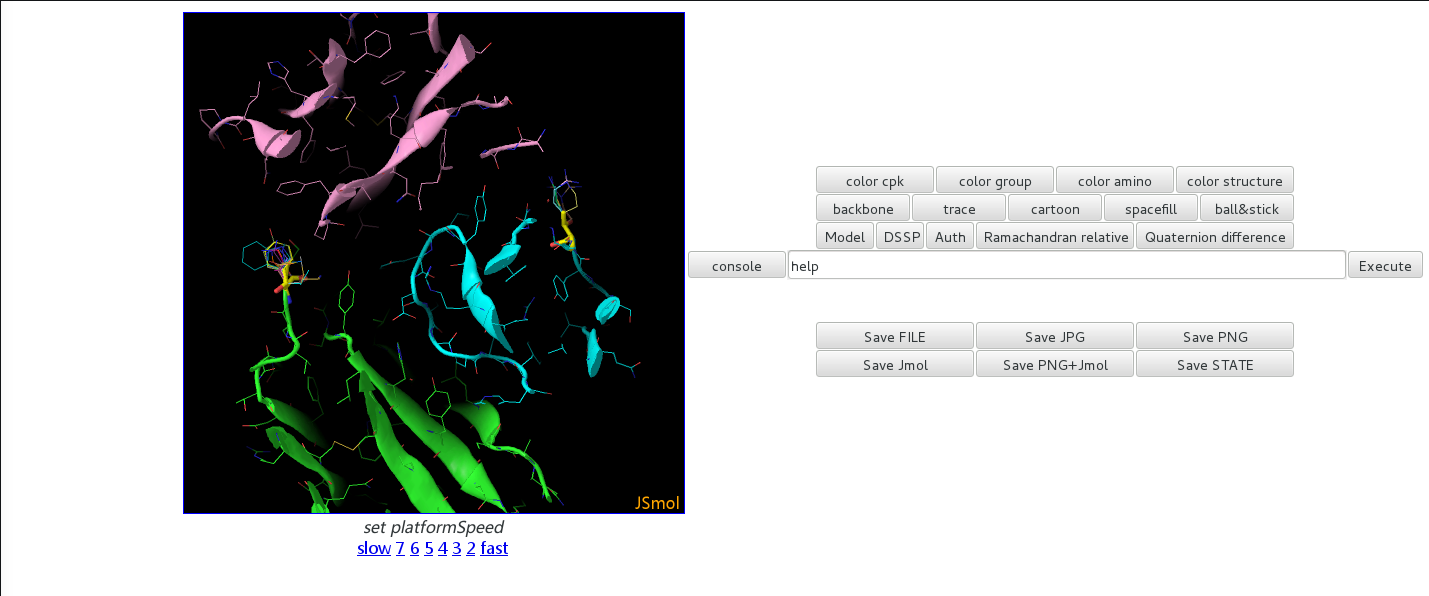

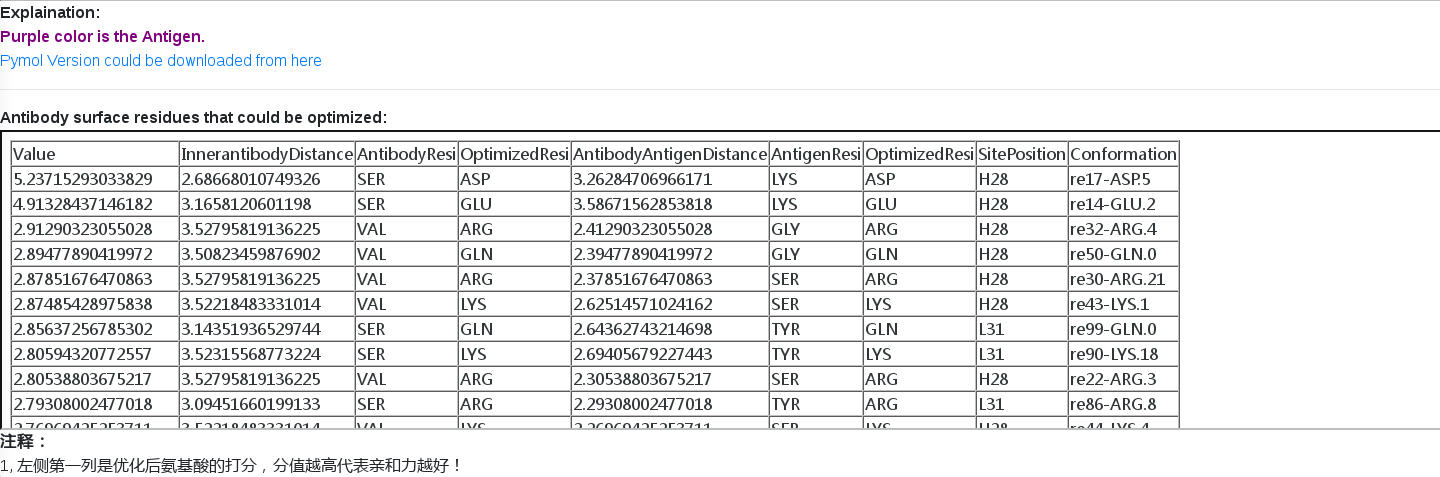
结果解读:
如上图所示:分为上下2个部分,上面的是结构的示意图,下面的是能量打分等统计值。
在上面部分的结构中我们可以看到,黄色的就是我们输入的位点,可以看到在这些黄色的位点位置上有各种其它的替换的氨基酸及其不同的构象结构,这些氨基酸的构象可以通过结构来观察。
下面的部分是一个表格形式,其中包含了上面结构显示的不同位置的各种氨基酸的分值。从左至右分别为:
Value:根据我们的算法,通过距离、作用力、电荷等等综合打分,分值越高亲和力越好。
InnerantibodyDistance:判断突变后的氨基酸是否与抗体内部其它的氨基酸有所冲突。
AntibodyResi:与优化突变后的氨基酸最近的抗体氨基酸。
OptimizedResi:优化突变后的抗体氨基酸。
AntibodyAntigenDistance:判断突变后的氨基酸与抗原氨基酸直接的距离。
AntigenResi:与优化突变后的氨基酸最近的抗原氨基酸。
OptimizedResi:优化突变后的抗体氨基酸。
SitePosition:优化突变的位置。
Conformation:优化突变后的氨基酸的构象结构,比如ASP.5就是谷氨酸的第5个构象。
通过上表的第一列的总体打分我们可以得出结论,在H链的第28位氨基酸的位置可以优化突变成D或者E来提高亲和力。